近年来,单细胞RNA测序技术(scRNA-seq)的快速发展为生命科学研究带来了革命性的变化。这项技术使科学家们能够在单个细胞水平上解析基因表达谱,为理解细胞异质性、发育轨迹和疾病机制提供了前所未有的分辨率。然而,在基因调控网络(GRN)研究领域,科学家们仍然面临着几个关键性挑战:
分辨率瓶颈:传统GRN推断方法(如GENIE3、GRNBoost2等)通常只能在细胞群体水平构建“平均化”的调控网络,无法反映单个细胞特有的调控特征。这种"群体平均"的方法掩盖了细胞间的重要异质性信息。
因果关系困境:现有方法主要基于基因间的共表达关系,难以区分真实的因果关系和相关关系。这种局限性使得研究人员无法准确识别调控关系中的"驱动者"和"响应者"。
动态过程解析不足:对于细胞重编程、发育分化等动态过程,传统方法缺乏时间维度的建模能力,难以捕捉调控关系的动态演变规律。
这些挑战严重制约了科学家们对复杂生物系统的深入理解,也阻碍了相关研究成果向临床应用的转化。
2025年6月27日,国科大杭州高等研究院(以下简称杭高院)生命与健康科学学院(以下简称生命学院)/上海交通大学陈洛南教授和刘小平教授作为共同通讯在Research(IF=10.7)在线发表题为 “Constructing Cell-Specific Causal Networks of Individual Cells for Depicting Dynamical Biological Processes”的研究论文。

该研究提出了一种名为SiCNet的新型单细胞因果网络构建方法。该方法通过整合单细胞转录组数据和因果推断策略,精准识别单个细胞中基因间的因果关系,突破了传统方法仅能分析细胞群体水平的局限,为解析细胞异质性、癌症进展和细胞重编程等复杂生物学过程提供了新工具(图1)。
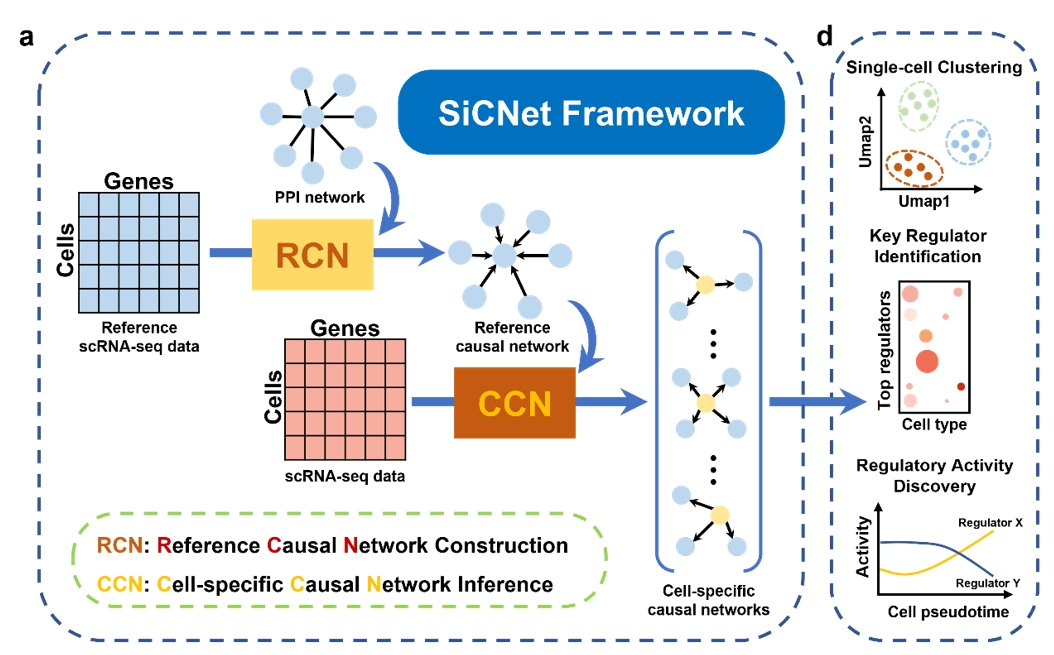
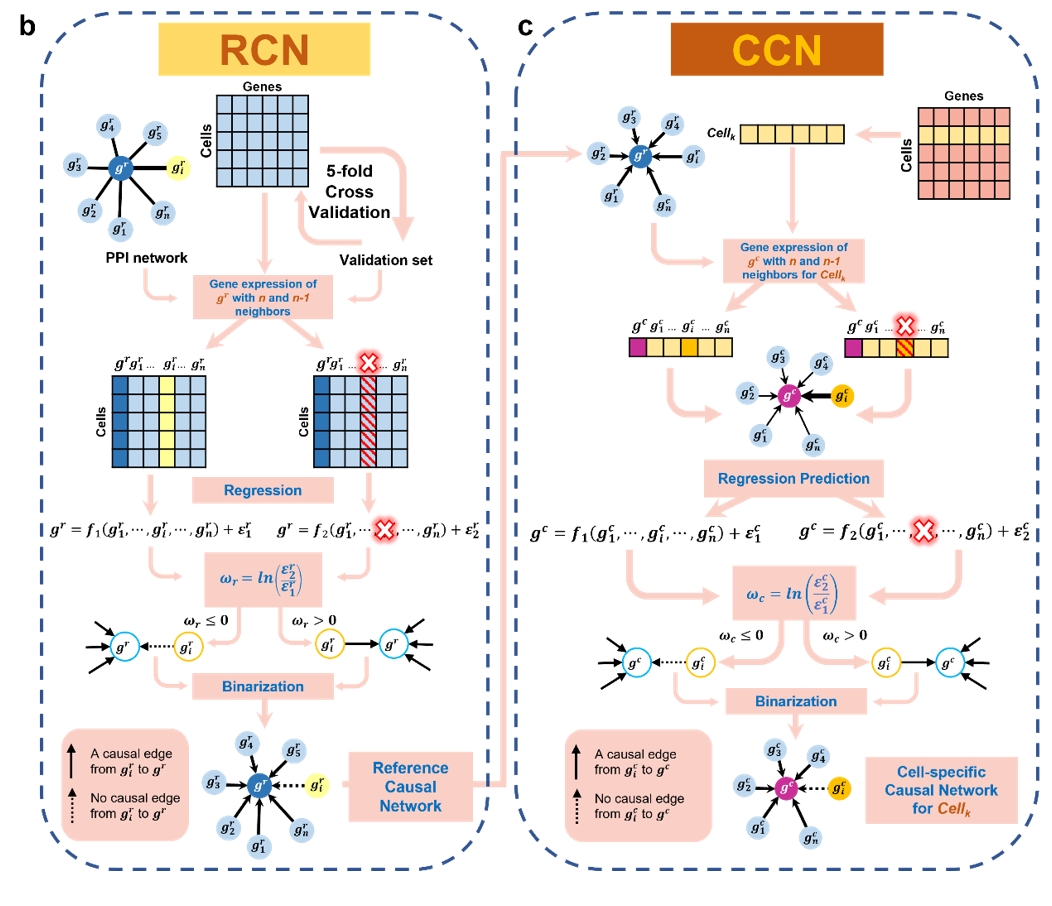
图1、SiCNet算法框架
在本研究中,研究者创新性地提出网络出度矩阵(ODM)的概念,将复杂的调控网络信息转化为数值矩阵。ODM的每个元素代表特定基因在单个细胞中的调控活性强度(即调控下游靶基因的数量)。这种转换使得网络特征可以直接应用于各种单细胞分析流程。通过分析6,168个结直肠癌的上皮细胞数据,涵盖健康组织区域、肿瘤交界区和肿瘤核心区。SiCNet鉴定出26个在肿瘤进展中调控活性显著增强的关键因子,包括ABL1、NCOA1等。此外,SiCNet发现133条显著上调的调控关系,如ABL1→RAC3等,这些关系与肿瘤细胞的化疗抗性和干性维持密切相关。通路分析显示这些因子富集在PPAR信号通路、胰岛素抵抗等代谢相关通路,为结直肠癌的代谢重编程提供了新证据。(图2)。
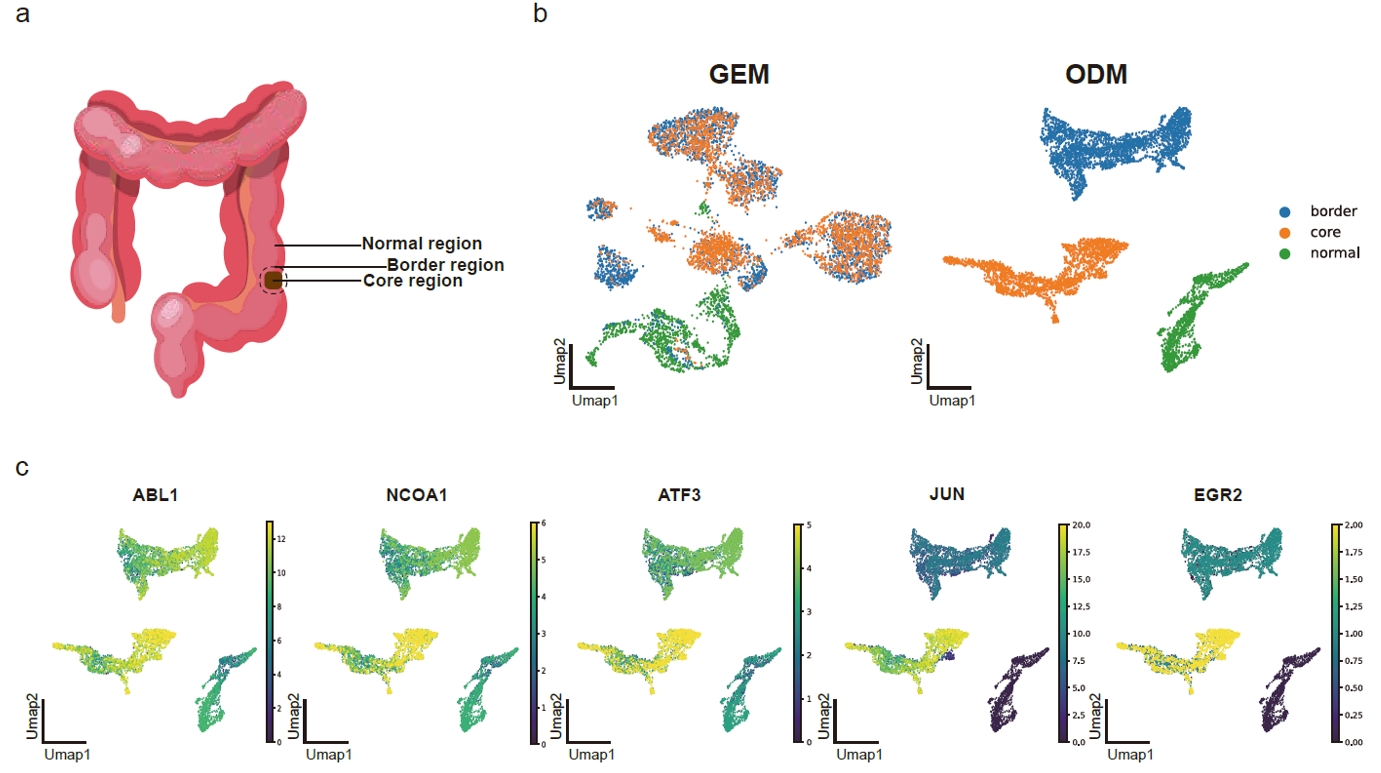
图2、结直肠癌中鉴定出的关键调控因子
此外,SiCNet还应用于细胞重编程和造血发育研究,揭示了动态调控网络中的关键基因及其作用机制。综上,这项研究报道的SiCNet能更精准识别生物过程中的关键调控基因,揭示动态过程中的调控机制。该方法不仅适用于单细胞转录组数据,还可扩展至空间转录组学等领域,为精准医学、药物开发和合成生物学研究提供了新工具。
国科大杭高院为该工作的第一单位。杭高院生命学院2022级博士研究生黄忻哲为本论文第一作者,陈洛南教授和刘小平教授为共同通讯作者。该工作得到了国家自然科学基金、中科院、杭高院基金等支持。
论文链接:https://spj.science.org/doi/10.34133/research.0743
Github链接:https://github.com/Huang-XZ-Sandy/SiCNet